(著)山たー
CourseraのComputational Neuroscienceの2週目。読む上での注意点はWeek0を参照してください。
主にエンコーディングについて
●脳から神経活動を記録する技術
●ニューロンの発火を起こす刺激特徴量の求め方
2.1:What is the Neural Code?
非侵襲的な脳活動測定
脳の活動を非侵襲的に計測する方法
●fMRI (functional Magentic Resonance Imaging; 機能的磁気共鳴画像法)
(利点)神経活動の領域を近似的に発見するのに役立つ
(欠点)時間分解能は悪い
●EEG (Electroencephalography; 脳波計測)
(利点)脳内の神経回路をより迅速に電気的に捕捉することができる(時間分解能が良い)
(欠点)空間分解能がfMRIより悪い。ノイズが多い
その他の計測法として、MEG(Magnetoencephalography; 脳磁図検査法), PET(Positron Emission Tomography;ポジトロンエミッション断層撮影法)などがある。 → 医用工学
これらの問題点は、個々のニューロンの活動を記録できないこと。
侵襲的な脳活動測定
●電極アレイ(Electrode arrays):
2mm程の電極を多数並べたもの。剣山みたいなかたちをしている。BMIによく使われる。
●カルシウムイメージング(calcium imaging)
Ca2+蛍光指示薬を用いて、細胞内のCa2+の濃度変化を画像的に計測する。
微小電極を直接イオンチャネルに押し当てて神経細胞の膜電位変化を直接計測する。電気信号の減衰により、外部から記録した電位変化は振幅が小さいため、かなり増幅する必要がある。細胞内部で記録することで、記録される信号の振幅は大きくなる。
※ただし、かなり技術が必要
Spike raster plot
ラスタープロット(raster plot)は、複数のニューロンの活動の時間変化を縦に並べたもの。次図は動画を見ているときのラスタープロット。
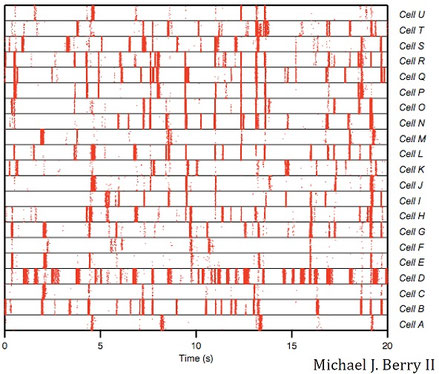
縦軸が細胞のラベル、横軸が時間となっている。赤色の縦線がニューロンの発火を表す。赤色が強く線が太い場合、その時の刺激に対してニューロンが強く反応することを示す。赤色が弱い場合、反応はするが、最もよく反応する刺激ではないということ。
縦に並んだ線は発火の一致を示す。つまり、発火タイミングが同じ場合、複数のニューロンが同じ刺激に対して発火したということである。ラスタープロットにより、各ニューロンを機能別に分類することができる。
EncodingとDecoding
Stimulus(刺激)とResponse(反応)はそれぞれ、$S$, $R$と略されることが多い。
刺激パラメータの選択
刺激に対する反応を考えるにあたり、刺激の中のどの部分がパラメータとなっているかを考えなければならない。つまり、刺激パラメータ(Stimulus parameter)を選択する必要がある。
例えば、刺激が特定の音節を話す人の音声録音である場合、刺激パラメータとして、次の2つが考えられる。
①信号の平均振幅
②信号中の2つの周波数成分の相対振幅
一般に、ニューロンの応答に影響を及ぼす可能性がある、異なった刺激パラメータは多数存在する。良い実験を設計するには、他の全てのパラメータをできるだけ一定に保ちながら調べるための自然なパラメータセットを選ばなければならない。皮質下領域のニューロンでは、どのパラメータが重要であるか(例えば、外側膝状体(LGN)のニューロンだと光のスポットの位置)を把握することは、比較的簡単である。しかし、より高次の皮質領域では、ニューロンがどの刺激パラメータによって調節されているかを正確に理解することはとても難しい。
同調曲線 (Tuning curves)

上図はV1のニューロンについての同調曲線。任意の傾きでも弱く反応するが、最も強く反応するのは特定の傾きのみである。
2.2:Neural Encoding: Simple Models
Encodingモデルの構築
線形フィルタモデル
●スカラー倍モデル
$r(t)$がスカラー倍によって求められる場合は $$ r(t)=\phi s(t) $$ のようになる。

変換に線形フィルタ(linear filter)を用いてみる。
①時系列フィルタモデル (temporal filtering model)
離散時間: $$r(t)=\sum_{k=0}^n s_{t-k}f_k$$ 連続時間: $$r(t)=\int_{-\infty}^t d\tau s(t-\tau)f(\tau)$$
数値計算上は離散時間の式を用いることとなる。刺激(ホワイトノイズ)を生成し、3つの一次元線形フィルタをかけると次図のようになる。1段目がWaveletFilter(の一部)、2段目が移動平均フィルタ(moving average filter)、3段目が指数移動平均フィルタ(exponential moving average filter; leaky filter)である。

import numpy as np
import matplotlib.pyplot as plt
from scipy import signal
t_len = 100
f_len = 10
#stimuli (white noise)
mean = 0
std = 1
s = np.random.normal(mean, std, size=t_len)
# wavelet filter
def WaveletFilter(start, f_len, t_len = 100, amp = 4.0):
f = np.zeros([t_len])
partial_f = signal.ricker(f_len*1.9, amp)
partial_f = partial_f[:f_len]
f[start:start+f_len] = partial_f
return f
# running average filter
def RunnningAverageFilter(start, f_len, t_len = 100):
f = (1*(t>start) - 1*(t>start+f_len))/f_len
return f
#Exponential Moving Average
def LeakyAverageFilter(start, f_len, t_len = 100):
f = np.zeros([t_len])
partial_f = np.exp(np.arange(-f_len,0, 1)+1)
f[start:start+f_len] = partial_f
return f
# running average filter
t = np.arange(0,t_len, 1)
# reaction
r_W = np.zeros([t_len])
r_RA = np.zeros([t_len])
r_LA = np.zeros([t_len])
for i in range(t_len-f_len):
f = WaveletFilter(i, f_len)
r_W[i] = np.sum(s*f)
for i in range(t_len-f_len):
f = RunnningAverageFilter(i, f_len)
r_RA[i] = np.sum(s*f)
for i in range(t_len-f_len):
f = LeakyAverageFilter(i, f_len)
r_LA[i] = np.sum(s*f)
# plot fig
plt.figure(figsize=(10,10))
plt.subplot(3,3,1)
plt.title('Stimuli: s(t)')
plt.xlabel("t")
plt.plot(s)
plt.subplot(3,3,2)
plt.title('Wavelet Filter: f')
plt.xlabel("k")
plt.plot(WaveletFilter(5, f_len, t_len=f_len*2))
plt.subplot(3,3,3)
plt.title('Reaction: r(t)')
plt.xlabel("t")
plt.plot(r_W)
plt.subplot(3,3,4)
plt.title('Stimuli: s(t)')
plt.xlabel("t")
plt.plot(s)
plt.subplot(3,3,5)
plt.title('Runnning Average Filter: f')
plt.xlabel("k")
plt.plot(RunnningAverageFilter(5, f_len)[:f_len*2])
plt.subplot(3,3,6)
plt.title('Reaction: r(t)')
plt.xlabel("t")
plt.plot(r_RA)
plt.subplot(3,3,7)
plt.title('Stimuli: s(t)')
plt.xlabel("t")
plt.plot(s)
plt.subplot(3,3,8)
plt.title('Leaky Average Filter: f')
plt.xlabel("k")
plt.plot(LeakyAverageFilter(5, f_len, t_len=f_len*2))
plt.subplot(3,3,9)
plt.title('Reaction: r(t)')
plt.xlabel("t")
plt.plot(r_LA)
plt.tight_layout()
plt.savefig("linear_filter.png")
plt.show()
②空間的フィルタモデル (spatial filtering model) \begin{align*} r(x,y)&=\sum_{x'=-n, y'=-n}^n s_{x-x',y-y'}f_{x',y'}\\ &=\int_{-\infty}^{\infty} dx'dy'\ s(x-x',y-y')f(x',y') \end{align*}

from skimage import data, color, filters, img_as_float
from matplotlib import pyplot as plt
original_image = img_as_float(data.camera())
img = color.rgb2gray(original_image)
sigma = 1.5
k = 2
s1 = filters.gaussian_filter(img,sigma)
s2 = filters.gaussian_filter(img,sigma*k)
dog = s1 - s2
plt.subplot(1,2,1)
plt.imshow(original_image)
plt.title('Original Image')
plt.subplot(1,2,2)
plt.imshow(dog,cmap='RdBu')
plt.title('DoG with sigma=' + str(sigma) + ', k=' + str(k))
plt.savefig("DoG.png")
plt.show()
③時空間的フィルタモデル (spatiotemporal filtering model) $$ r_{x,y}(t)=\int\int\int dx'dy'd\tau f(x',y',\tau)s(x-x',y-y',t-\tau) $$
時空間ガボールフィルタなど。Neural Networkにおける3D convolutionのフィルタも同じ。エンコーディングでは結構重要で、例えば西本伸志先生の運動エネルギーモデルは、時空間ガボールフィルタを使っている。 → gallantlab/motion_energy_matlab
※西本先生の話はWeek3に出てくる。
2.3:Neural Encoding: Feature Selection
線形フィルタと非線形関数の組み合わせ
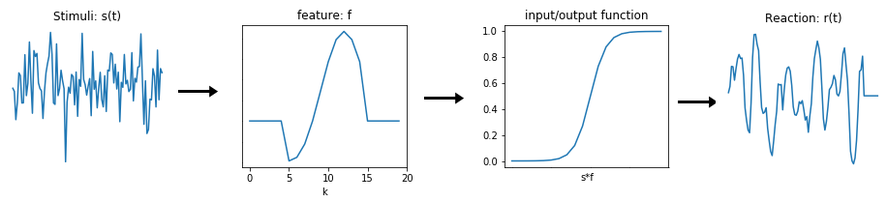
import numpy as np
import matplotlib.pyplot as plt
from scipy import signal
t_len = 100
f_len = 10
t = np.arange(0,t_len, 1)
#stimuli (white noise)
mean = 0
std = 1
s = np.random.normal(mean, std, size=t_len)
# wavelet filter
def WaveletFilter(start, f_len, t_len = 100, amp = 4.0):
f = np.zeros([t_len])
partial_f = signal.ricker(f_len*1.5, amp)
partial_f = partial_f[:f_len]
f[start:start+f_len] = partial_f
return f
def Sigmoid(x):
return 1/(1+np.exp(-x))
# reaction
r = np.zeros([t_len])
for i in range(t_len-f_len):
f = WaveletFilter(i, f_len)
r[i] = np.sum(s*f)
r = Sigmoid(r)
# plot fig
plt.figure(figsize=(12,3))
plt.subplot(1,4,1)
plt.title('Stimuli: s(t)')
plt.xlabel("t")
plt.plot(s)
plt.subplot(1,4,2)
plt.title('feature: f')
plt.xlabel("k")
plt.plot(WaveletFilter(5, f_len, t_len=f_len*2))
plt.subplot(1,4,3)
plt.title('input/output function')
plt.xlabel("s*f")
plt.plot(Sigmoid(np.arange(-10,10, 1)))
plt.subplot(1,4,4)
plt.title('Reaction: r(t)')
plt.xlabel("t")
plt.plot(r)
plt.tight_layout()
plt.savefig("LinearFliterAndNonlinearFunction.png")
plt.show()
ここで問題となるのは、どのようにして特徴量を取り出すかである。
Gaussian white noiseを用いて特徴量を得る
連続な刺激$s(t)$を離散化して、特徴量$s_1, s_2, \cdots, s_n$によって成り立っているとする。つまり、 $$ s(t)\approx \{s_1, s_2, \cdots, s_n\} $$ とする。


スパイクトリガー平均 (Spike Triggered Average)
スパイクを誘発する刺激を平均化したものをスパイクトリガー平均(spike-triggered average; STA)という。

3次元データ(x, 画素値, 時間)なら次のようになる。

(引用元)
Schwartz, O, Pillow, JW, Rust, NC, and Simoncelli, EP. (2006). Spike-triggered neural characterization. Journal of Vision, 6(4):484-507
線形フィルタの意味
線形フィルタ = 畳み込み =(高次元刺激空間への)投影
非線形の入力/出力関数(活性化関数)の決定
複数の特徴量を用いる場合

主成分分析で高次特徴量を抽出する
ニューロンが高次元の特徴量に応答する場合、単に加算平均を取るSTAではうまくいかない場合がある。そのような時、主成分分析(principal component analysis; PCA)を行う。
例えば特定の顔に反応するニューロンがあったとして、顔のどのような成分が刺激に寄与しているのか調べたいとする。このような場合、PCAを用いて、固有顔(Eigenface)を生成するという方法がある。固有顔は、顔の主成分のうち寄与率が高いものを選択したものである。
ここで、Pythonで固有顔を求めてみる。データセットはOlivetti facesを使う。まず元の顔画像(の一部)。

PCAをかけた結果。画像の上の数字は寄与率。

単に加算平均をとると、次のようになる。

import numpy as np
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import fetch_olivetti_faces
"""
load Olivetti faces dataset
oliv.images (400, 64, 64) : 400 face images
oliv.data (400, 4096) : reshape images (4096=64x64)
oliv.target (400,) : 40 label of faces
"""
oliv = fetch_olivetti_faces()
# Show 64samples
fig = plt.figure(figsize=(6,6))
fig.subplots_adjust(left=0, right=1, bottom=0, top=1, hspace=0.05, wspace=0.05)
for i in range(64):
ax = fig.add_subplot(8, 8, i+1, xticks=[], yticks=[])
ax.imshow(oliv.images[i], cmap=plt.cm.bone, interpolation='nearest')
#plt.savefig("fetch_olivetti_faces.png")
plt.show()
plt.close()
# Input data
X, y = oliv.data, oliv.target
# Eigen Face
num_people = 4 #人物数
n_comp = 5 #次元数
fig = plt.figure(figsize=(6,6))
plt.title("Eigen Faces")
plt.axis("off")
for i in range(num_people):
# Compute Principle Component for each person
face_data = X[y==i]
pca = PCA(n_components=n_comp)
pca.fit(face_data)
components = pca.components_
E = pca.explained_variance_ratio_ #寄与率
# Show result
for j in range(n_comp):
ax = fig.add_subplot(num_people, n_comp, 1 + n_comp * i + j,
xticks=[], yticks=[])
ax.set_title("{0:01.3f}".format(E[j]))
if j==0:
ax.set_ylabel("No."+str(i), rotation=0, fontsize=10, labelpad=20)
ax.imshow(components[j, :].reshape(64, 64))
plt.tight_layout()
plt.savefig("EigenFaces.png")
plt.show()
plt.close()
# Average Face
fig = plt.figure(figsize=(6,2))
plt.title("Average Faces")
plt.axis("off")
for i in range(num_people):
# Compute Principle Component for each person
face_data = X[y==i]
ave_face = np.average(face_data, axis=0)
ave_face = np.reshape(ave_face, (64,64))
# Show
ax = fig.add_subplot(1, num_people, i+1,
xticks=[], yticks=[])
ax.set_title("No."+str(i))
ax.imshow(ave_face)
plt.tight_layout()
plt.savefig("AverageFaces.png")
plt.show()
STAはスパイクを引き起こす刺激の単純な特徴を与える。しかし、STAは単純に加算平均をするだけなので、一部の興味深いダイナミクスをとらえることができない。例えば、ニューロンが+から-への変化と-から+への変化、両方に反応する場合、スパイクトリガー刺激は平均してゼロになり、STAは平らな線のようになってしまう。これは全く有用ではない。
別の言葉で言えば、STAは分散情報が失われる。PCAを使うことで、刺激データの分散を保持することができる。また、PCAにより、ニューロンが求める刺激の特徴を表すのに必要な次元数を求めることができる。
PCAを用いる他の例としてSpike sortingがある。
2.4:Neural Encoding: Variability
Kullback-Leibler divergenceを用いた特徴量選択
スパイクが刺激特異的に生じる場合に意味がある。もし、刺激とスパイクの発生が独立していれば、それは何の意味もない。

import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import norm
x_axis = np.arange(-5, 5, 0.001)
p_s = norm.pdf(x_axis,0,1)
p_s_spike1 = norm.pdf(x_axis,0.2,1)
p_s_spike2 = norm.pdf(x_axis,2,0.5)
plt.figure(figsize=(9,4.5))
plt.subplot(1,2,1,xticks=[], yticks=[])
plt.plot(x_axis, p_s, label="p(sf)")
plt.plot(x_axis, p_s_spike1,label="p(sf|spike)")
plt.title("Boring:\n spikes unrelated to stimulus")
plt.xlabel("sf", fontsize=20)
plt.legend(loc='upper left')
plt.subplot(1,2,2,xticks=[], yticks=[])
plt.plot(x_axis, p_s, label="p(sf)")
plt.plot(x_axis, p_s_spike2,label="p(sf|spike)")
plt.title("Interesting:\n spikes are selective")
plt.xlabel("sf", fontsize=20)
plt.legend(loc='best')
plt.tight_layout()
plt.savefig("spike_gaussain.png")
この場合、刺激$s_f$の分布$P(s_f)$と、スパイクが発生する下での$s_f$の分布$P(s_f|\text{spike})$が異なる場合、$s_f$は意味のある特徴量であるといえる。つまり、$P(s_f)$と$P(s_f|\text{spike})$の「距離」が大きければよいわけである。
ここで、分布の距離の尺度としてKullback-Leibler divergence $(D_{KL})$がある。$D_{KL}$は次のように計算できる。 $$ D_{KL}(P(s),Q(s)) = \int ds P(s) \log_2 \frac{P(s)}{Q(s)} $$ 今の場合、$D_{KL}(P(s_f|\text{spike}), P(s_f))$を求め、この$D_{KL}$を最大化するフィルターを選べばよい。

これは刺激とスパイクの相互情報量を最大化するのと同じである。この手法を用いれば、ホワイトノイズ入力に依存せず、自然な刺激からモデルを導出することができる。
まとめると、ある特徴量と関連する特徴量を探すには次のようにする。
1.条件付き期待値(平均)を用いて単一のフィルタを求める。
2.PCAを用いて得られるフィルタの組を用意する
3.情報理論的手法(KL最大化)を用いる
こうすることでホワイトノイズ刺激を用いなくてもよくなる。
スパイクが二項分布に従う場合
スパイクがポアソン分布に従う場合
次にポアソン分布を考える。ポアソン分布は次図のような分布。

import numpy as np
from scipy.stats import poisson
from matplotlib import pyplot as plt
mu_values = [1, 5, 15]
x = np.arange(-1, 30)
plt.figure(figsize=(5,4))
for mu in mu_values:
plt.plot(x, poisson.pmf(x,mu), label="μ = "+str(mu))
plt.xlim(-0.5, 30)
plt.ylim(0, 0.4)
plt.xlabel("x")
plt.ylabel("p(x|μ)")
plt.title("Poisson Distribution")
plt.legend()
plt.savefig("PoissonDistribution.png")
plt.show()
スパイクの発火率(firing rate)を$r$としたとき、発火が$T$秒間に$k$回起こる場合、 $$P_T(k)=(rT)^k \frac{\exp(-rT)}{k!}$$ となる。
例えば、1秒間に4回のスパイク$(r=4 \text{spikes/second})$の割合で発火。ポアソンスパイクの発火が$T$秒間に$k$回発火するとすると、スパイクは $$p(k) = (rT)^k \frac{e^{-rT}}{k!}$$ に従う。1秒間発火しない確率は $$p(0)=1\cdot \frac{e^{-4}}{0!}=e^{-4}$$ と計算できる。
ポアソンスパイクは、各スパイクの生じた時刻が他のスパイクとは無関係であると仮定している。しかし、実際のニューロンでは、直前のスパイクが生じた直後にニューロンが発火するのを防ぐ不応期(通常は1ミリ秒程度)が存在する。1秒あたりのニューロンのスパイク数が多いほど、この不応期の影響は大きくなる。
さらに、ニューロンの発火をポアソン分布に従わなくさせる、より複雑な影響もある。端的に言えば、他のニューロンとの相互作用である。ゆえに、あるニューロンのスパイク発生モデルには、そのニューロン自体の発火の履歴と、他のニューロンの相互作用を考慮にいれなくてはならない。
一般化線形モデル
上記のことを踏まえたモデルとして、一般化線形モデル(Generalized linear model; GLM)を用いた次のようなモデルがある。
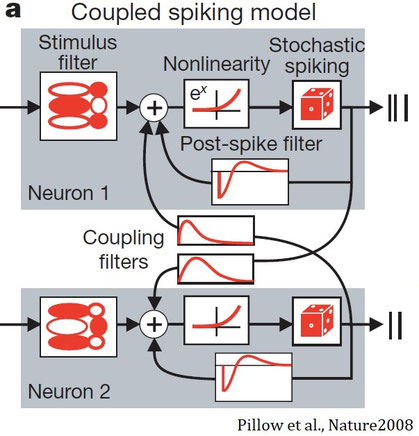
主要な部分はLinear-nonlinear-Poisson cascade modelである。これは
刺激フィルタ→非線形→確率的スパイク
と変換するモデルである。上図のモデルでは、さらにPost spike filter(自身へのフィードバック)とCoupling filters(他のニューロンへのフィードバック)を加えている。式としては次のようになる。
Programming Quiz
長くなるので別記事にしました。 → スパイクトリガー平均・共分散の計算法 (Python)
コメントをお書きください
佐藤 亜子 (月曜日, 28 6月 2021 08:53)
Michael J Berry のグラフはどの論文からの引用でしょうか。ある番組で使用したいのですが、著作権フリーのもでしょうか。それともコモンクリエイエィティブライセンスでしょうか。このメルアドにお返事いただければ幸いです。
a.sato@amalate.co.jp
gemmalyly (月曜日, 24 10月 2022 17:53)
非常に有益で有益な投稿、記事をありがとう。 休憩時間にhttps://nerdle-game.coに立ち寄って最高のゲームをプレイすることもできます